
Já falamos aqui no blog da necessidade de se fazer as perguntas certas em projetos de dados e dos desafios da comunicação do trabalho remoto.
Um outro aspecto mais técnico que faz parte do dia-a-dia dos cientistas de dados que vamos abordar hoje é a importância de garantir que todo código criado em um projeto de dados seja acessível e reprodutível por todos os membros da equipe de cientistas de dados.
Isso significa que cada cientista precisa instalar todas as bibliotecas e dependências de software em sua própria máquina para ser capaz de reproduzir os algoritmos e análises uns dos outros. Se não houver uma estratégia organizada, há o risco de que uma pessoa instale uma versão diferente de alguma biblioteca (ex: numpy e pandas incompatíveis) que quebre todo o código!
Você já deve ter ouvido ou dito a frase "Não sei porque não funciona na sua máquina, na minha funcionou normal!". Pois é, o Docker existe para evitar que isso aconteça.
Primeiro: o que é Docker?
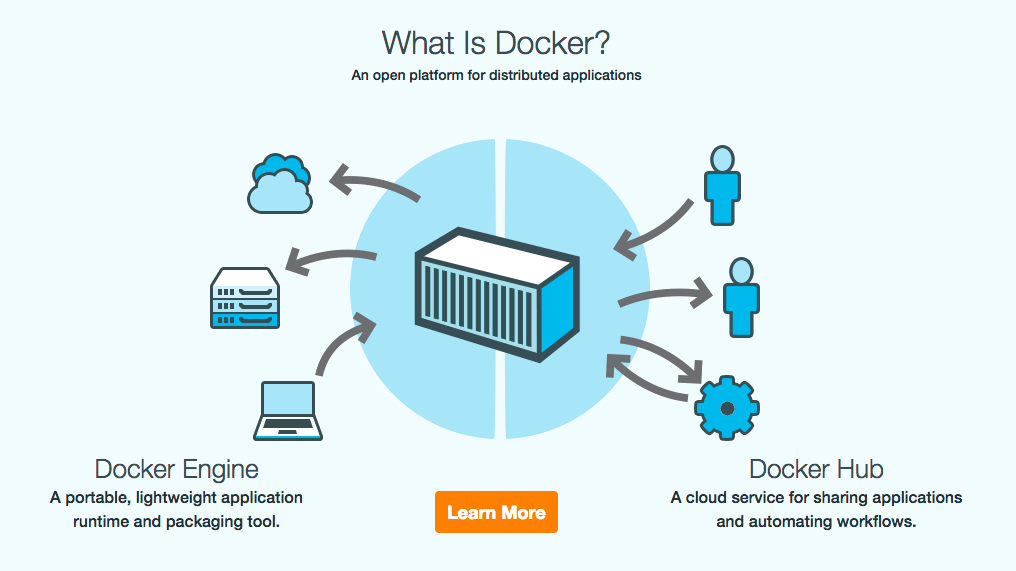
O Docker é a ferramenta mais conhecida e usada atualmente de software de contêineres. Ele permite por meio da virtualização a nível de sistema operacional transformar um conjunto de pacotes diferentes em uma coisa só. Esses contêneres são isolados e podem se comunicar entre si por canais bem definidos.
Quando começamos um projeto de ciência de dados na DSB, quase sempre precisamos dos seguintes elementos:
- a possibilidade de executar scripts Python ou bash
- um banco de dados simples só para armazenar alguns resultados
- um servidor Spark standalone
Imagine ter que garantir que em cada computador, cada ambiente, todos os pacotes python, jars e afins precisem ser os mesmos... E se parte da equipe usa Ubuntu, outra parte MacOS e uma terceira Windows? Como garantir que a aplicação vai rodar da mesma forma em todos esses ambientes? Se o cliente que vai usar esse código depois possuir um servidor em Windows e a equipe usar Linux, como garantir a integridade da solução?
O Docker resolve esses problemas ao garantir que todos trabalhem exatamente com o mesmo ambiente, mesmas bibliotecas, mesmos serviços e isso sem que se precise instalar manualmente todos os pacotes e linguagens de programação.
Com o Docker, se alguém da equipe desenvolver uma solução nova, basta adicionar as linhas de código necessárias aos contêineres do projeto que essa solução pode ser replicada no ambiente de toda equipe.
Os principais conceitos envolvidos
Para entender como o Docker permite essas facilidades citadas acima, precisamos nos ambientar com alguns termos e conceitos importantes da ferramenta:
Contêiner:
Pense no contêiner como uma máquina virtual que roda no seu computador. O contêiner é uma unidade de software que une todo o código e suas dependências para que a aplicação funcione consistentemente e de forma rápida independente do ambiente de execução. Quando tiver rodando aplicações no Docker, você pode conferir quais contêineres estão ativos com o comando docker stats.
Os contêineres são definidos a partir de uma Imagem.
Imagem:
Uma imagem Docker é um arquivo que define como um contêiner será criado. É como se fosse um template para se criar uma máquina virtual.
Ela é construída a partir de uma série de instruções definidas por meio do arquivo de texto Dockerfile. Nele, deverá constar todas as dependências, instalações de softwares e pacotes de linguagens de programação, enfim tudo o que é necessário para uma aplicação que será executada usando o kernel da máquina de host. Quando uma imagem é instanciada, ela cria um Contêiner.
Como exemplo, o Dockefile abaixo é o que utilizamos para executar Jupyter notebooks nos nossos projetos:
# Jupyter pyspark image from https://hub.docker.com/r/datasciencebrigade/jupyter-pyspark-image
FROM datasciencebrigade/jupyter-pyspark-image:1.2
USER jovyan
WORKDIR /mnt/code
COPY requirements.txt requirements.txt
RUN pip install --upgrade pip
RUN pip install -r requirements.txt
EXPOSE 8888O arquivo começa com o comando FROM, indicando que esta imagem herda de uma outra imagem pai - que está disponível no Docker Hub - baseada no Ubuntu e que já tem todas as dependências de Python instaladas. O arquivo completo você pode conferir no nosso Github.
Por padrão, gostamos de usar o /mnt/code como o diretório padrão (WORKDIR) dentro do Docker. Assim, sempre que acessarmos o shell do contêiner, iremos entrar por padrão nesse diretório.
Para copiar arquivos da máquina local para dentro do contêiner, é necessário usar o comando COPY. No exemplo acima, copiamos o arquivo requirements.txt que define as bibliotecas Python que precisamos para as nossas análises.
Um arquivo requirements.txt tipicamente define bibliotecas de análise de dados, visualizações, NLP, etc. Exemplo:
pandas==1.0.0
numpy
plotnine
matplotlib
fasttext==0.9.2
gensim>=3.0.0,<4.0.0Os trechos do Dockerfile com o comando RUN definem instruções a serem executadas no shell do contêiner. No exemplo acima, há comandos que instalam dependências de bibliotecas Python usando pip install.
Portas para acessar serviços dentro do contêiner são definidos no Dockerfile pelo comando EXPOSE.
Build & Run:
Para usar esse Dockerfile, você precisará primeiro construir essa imagem - e eventualmente lidar com erros caso tenha esquecido de definir a instalação de alguma dependência de código necessária. Para isso, use o comando docker build, atribuindo um nome para essa imagem. Exemplo:
docker build --tag ds-exploratory:latest .Pronto. Agora a imagem ds-exploratory pode ser utilizada para rodar um contêiner. Faça isso com o comando docker run:
docker run --name ds-exploratory-container ds-exploratoryAo final, você deverá ver algo do tipo:
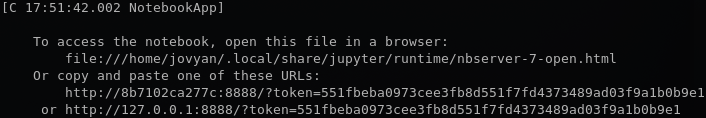
Cole essa URL que aparece no terminal (junto com o token) no navegador e pronto, você já poderá usar o Jupyter notebook que está rodando dentro desse contêiner Docker.
A imagem ds-exploratory que criamos com o comando build dá origem ao contêiner que chamamos de ds-exploratory-container.
Se quiser acessar o shell desse contêiner, use o comando docker exec. Deixe o contêiner rodando, abra um outro terminal e digite:
docker exec -ti ds-exploratory-container /bin/bashO parâmetro -ti te permite acessar o shell e digitar comandos de forma dinâmica.
Para sair da shell, digite exit.
Para parar o contêiner original, dê um Ctrl+C na tela do terminal onde está rodando.
Volume:
O contêiner que criamos acima roda em um ambiente isolado, todo arquivo que for criado lá dentro só fica visível dentro do próprio contêiner. Ao parar ou matar o contêiner, nenhum arquivo ficará visível "do lado de fora". É claro que isso não é prático sempre já que precisamos que os notebooks e outros códigos e arquivos continuem existindo mesmo depois que a gente fechar o contêiner.
Para manter os dados e arquivos, mapeie um diretório dentro do contêiner para apontar para algum outro diretório da sua máquina. Assim, tudo que for feito nessa pasta, permanecerá após o fim da vida do contêiner.
No Docker, faça isso com o parâmetro -v. Por exemplo, para mapear uma pasta local com dados do projeto (/home/user/project/data) para dentro do contêiner no diretório /mnt/data, faça o seguinte:
docker run --name ds-exploratory-container -v /home/user/project/data:/mnt/data ds-exploratoryRedes:
É possível também criar uma rede de contêineres para que os mesmo se conectem entre si. Por padrão a rede em que os contêineres sobem é a bridge
Docker-compose: gerenciamento fácil de imagens e contêineres
Como já deu para perceber, para subir contêineres na mão usando docker "puro", além de instanciar as imagens Docker que servirão de templates, precisamos também muitas vezes expor portas de serviços necessárias (ex: 5000 para um servidor HTTP, 8888 para o Jupyter, 4040 ou 7077 para Spark, etc.), montar volumes de dados e passar variáveis de ambiente para os contêineres.
À medida que o projeto vai se tornando mais complexo, se torna necessário também mapear compartilhar variáveis de ambiente entre vários contêineres, mapear dependência entre serviços diferentes e por consequência, os comandos vão se tornando mais complicados.
docker run -v /home/dsb/dados:/foo -w /foo -i -t --expose 80 -e MYVAR1 --env MYVAR2=foo --env-file ./env.list -itd --network=my-net ubuntu bashUma ferramenta muito útil para gerenciar melhor a execução dos contêineres e lidar com essa complexidade natural dos projetos é o Docker Compose. Essa ferramenta te livra de usar o comando acima e facilita a definição e executação de múltiplos contêineres Docker usando para isso apenas um arquivo: o docker-compose.yml.
Segue abaixo o exemplo de configuração de um arquivo docker-compose.yml que usamos em projetos de data science que envolvem tanto análises exploratórias (Jupyter notebooks) bem como módulos e pacotes Python compartilhados:
version: "3.5"
services:
research:
build:
context: research
args:
JUPYTER_EXTERNAL_PORT: ${JUPYTER_EXTERNAL_PORT}
volumes:
- ./research:/mnt/code
- ${DATA_VOLUME}:/mnt/data
environment:
TIMEZONE: America/Sao_Paulo
SPARK_PUBLIC_DNS: ${SPARK_PUBLIC_DNS}
SPARK_MASTER_PORT: ${SPARK_MASTER_PORT}
SPARK_WORKER_WEBUI_PORT: ${SPARK_WORKER_PORT}
ports:
- ${SPARK_WORKER_PORT}:8081
- ${SPARK_APPLICATION_PORT}:4040
- ${JUPYTER_EXTERNAL_PORT}:8888
- ${SPARK_MASTER_WEBUI_PORT}:8080
- ${SPARK_MASTER_PORT}:7077
image: research
container_name: research-${CONTAINER_TAG}
processing:
build: processing
env_file:
- .env
environment:
TIMEZONE: America/Sao_Paulo
SPARK_CONF_DIR: /conf
SPARK_PUBLIC_DNS: ${SPARK_PUBLIC_DNS}
SPARK_MASTER_PORT: ${SPARK_MASTER_PORT}
SPARK_WORKER_WEBUI_PORT: ${SPARK_WORKER_PORT}
SPARK_PUBLIC_DNS: ${SPARK_PUBLIC_DNS}
volumes:
- ./processing:/mnt/code
- ${DATA_VOLUME}:/mnt/data
- ./processing/conf/worker:/conf
image: processing
container_name: processing-${CONTAINER_TAG}
Neste arquivo dois contêineres são declarados: research e processing. Neles são definidos volumes, portas que serão mapeadas, variáveis de ambiente (environment), além de um arquivos com algumas variáveis de ambiente, além da imagem que será usada pelo build e o nome que será gerado para o contêiner.
Repare que existem duas formas de declarar variáveis de ambiente, no arquivo .env ou direto, dentro da seção environment. As variáveis definidas no .env ficam disponíveis dentro do contêiner - acessadas usando a sintaxe ${} - e podem ser utilizadas também para redefinir outras variáveis como por exemplo o uso de ${CONTAINER_TAG} acima para personalizar o nome dos contêineres.
Com isso, ao invés de executar os comandos com docker build e docker run cheios de parâmetros, é possível compilar e subir os dois contêineres definidos acima simultaneamente com os comandos:
docker-compose build
docker-compose upÉ possível também rodar somente uma das aplicações:
docker-compose up researchVale a pena conferir a documentação completa com todas as capacidades da ferramenta no site do Docker Compose.
Templates para projetos de dados
No nosso Github, você pode encontrar templates de estrutura Docker que usamos como ponto de partida nos nossos projetos de ciência de dados, tanto de imagens Dockers que adaptamos para nosso uso quanto estrutura de diretórios para uso nos projetos:
- dsb-images: https://github.com/Data-Science-Brigade/dsb-images
- dsb-templates: https://github.com/Data-Science-Brigade/dsb-templates
Vantagens do Docker

Resumindo, existem várias vantagens de se usar Docker em um projeto de ciência de dados:
- Portabilidade: É fácil reproduzir e portar a solução desenvolvida por uma pessoa para outros dentro da equipe.
- Deploy simplificado: Ao portar uma solução para o ambiente de produção, não será preciso instalar bibliotecas, pacotes de software, etc. Geralmente, basta configurar as variáveis do ambiente
- Consistência: Garantia de que o código do modelo ou script funcione exatamente da mesma forma independente de sistema operacional que está sendo utilizado.
- Independência de componentes: É fácil substituir partes de uma solução por outras ferramentas. Por exemplo,

